| 5′-Capping |
Modifikation am 5′-Ende der mRNA zur Stabilisierung und Erkennung durch Ribosomen. |
| Adenin (A) |
Stickstoffhaltige Base der DNA, paart mit Thymin. |
| Allele |
Varianten eines Gens an einer bestimmten Stelle auf dem Chromosom. |
| Aminosäure |
Baustein von Proteinen, codiert durch Codons. |
| Annealing |
Schritt der PCR, bei dem Primer an die DNA binden. |
| Antikörper |
Proteine des Immunsystems zur Erkennung von Antigenen. |
| Antiparallel |
Gegenläufige Orientierung der DNA-Stränge. |
| Autosomal |
Gen liegt auf einem der nicht-geschlechtsgebundenen Chromosomen. |
| Barr-Körperchen |
Inaktiviertes X-Chromosom in weiblichen Zellen. |
| Basen |
Adenin, Thymin, Guanin, Cytosin (Uracil in RNA). |
| Basen-Exzisionsreparatur |
Reparatur einzelner Basen in der DNA. |
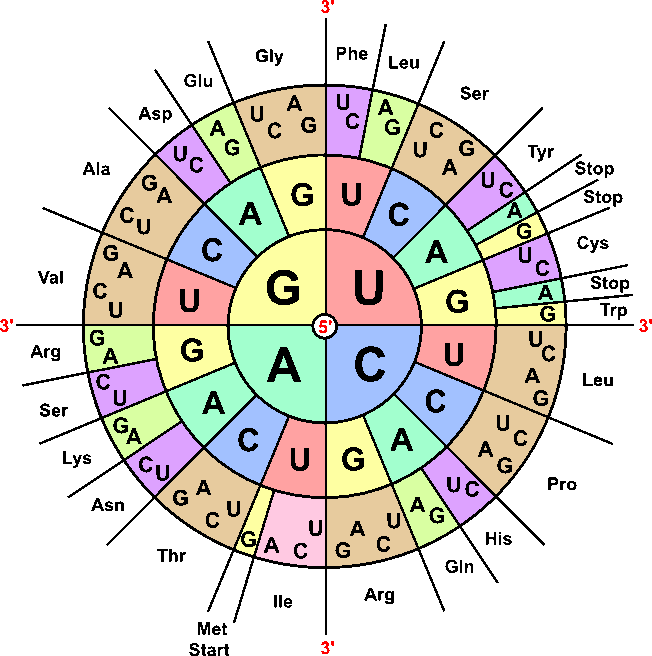
| Basentriplett / Codon |
Drei Basen, die eine Aminosäure codieren. |
| CRISPR/Cas9 |
Gentechnische Methode zur gezielten DNA-Veränderung. |
| Chiasma |
Sichtbare Überkreuzung homologer Chromosomen beim Crossing-over. |
| Chromatid |
Eine der beiden Hälften eines replizierten Chromosoms. |
| Chromatin |
Komplex aus DNA und Proteinen im Zellkern. |
| Chromosomen |
Strukturen aus DNA, die Gene enthalten. |
| Codogener Strang |
Der DNA-Strang, der bei der Transkription als Vorlage für die mRNA dient. |
| Crossing-over |
Austausch von DNA-Abschnitten während der Meiose. |
| DNA |
Desoxyribonukleinsäure, Träger der Erbinformation. |
| DNA-Methylierung |
Epigenetische Modifikation zur Genregulation. |
| DNA-Polymerase |
Enzym zur DNA-Synthese. |
| DNA-Reparaturmechanismen |
Systeme zur Erkennung und Behebung von DNA-Schäden. |
| Desoxyribose |
Zuckerkomponente der DNA-Nukleotide. |
| Dominant |
Ein Allel, das sich auch im heterozygoten Zustand phänotypisch ausprägt. |
| Dominanter Erbgang |
Ein dominantes Allel reicht zur Ausprägung eines Merkmals. |
| Enhancer/Silencer |
DNA-Abschnitte zur Steuerung der Genexpression. |
| Epigenetik |
Genregulation ohne Änderung der DNA-Sequenz. |
| Epistasie |
Ein Gen unterdrückt die Wirkung eines anderen (z. B. bei Albinismus). |
| Exom |
Alle codierenden Bereiche eines Genoms. |
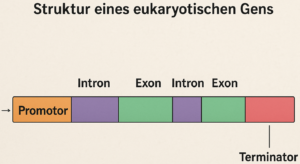
| Exon |
Codierender Abschnitt eines Gens. |
| Forensik |
Anwendung genetischer Verfahren zur Aufklärung von Straftaten. |
| Gameten |
Geschlechtszellen (Eizelle oder Spermium), haploid. |
| Gen |
Abschnitt der DNA, der für ein Protein codiert. |
| Genetischer Code |
Regelwerk zur Übersetzung von Basen in Aminosäuren. |
| Genkopplung |
Gene liegen nahe beieinander auf demselben Chromosom und werden gemeinsam vererbt. |
| Genmutation |
Veränderung einzelner Gene. |
| Genotyp |
Die genetische Ausstattung eines Organismus, z. B. AA, Aa, aa. |
| Genwirkkette |
Abfolge von Genen in einem Stoffwechselweg. |
| Grüne Gentechnik |
Gentechnik im Bereich der Landwirtschaft (z. B. transgene Pflanzen). |
| HDR |
Homologiegerichtete Reparatur, präzise Methode nach CRISPR/Cas9-Schnitt. |
| Helicase |
Enzym, das DNA-Stränge trennt. |
| Heterozygot |
Mischerbig – zwei unterschiedliche Allele (z. B. Aa). |
| Histonmodifikation |
Veränderung von Proteinen zur DNA-Verpackung. |
| Homozygot |
Reinerbig – beide Allele eines Gens sind gleich (z. B. AA oder aa). |
| Hybridisierung |
Bindung komplementärer DNA- oder RNA-Stränge – z. B. bei Sonden. |
| Initiation |
Beginn von Transkription oder Translation. |
| Interchromosomale Rekombination |
Neukombination durch zufällige Verteilung der Chromosomen. |
| Intermediärer Erbgang |
Erbgang, bei dem Heterozygote eine Mischform beider Merkmale zeigen. |
| Intrachromosomale Rekombination |
Neukombination durch Crossing-over innerhalb eines Chromosomenpaares. |
| Intron |
Nicht-codierender DNA-Abschnitt, wird ausgespleißt. |
| Karyogramm |
Chromosomen-Darstellung einer Zelle. |
| Keimbahn |
Zelllinie, aus der Gameten entstehen – genetisch relevant für Nachkommen. |
| Kettenabbruchmethode |
DNA-Sequenzierung nach Sanger durch Einbau von Abbruchnukleotiden. |
| Kodominanter Erbgang |
Beide Allele eines Gens sind gleich stark wirksam (z. B. Blutgruppe AB). |
| Kodominanz |
Beide Allele werden gleich stark exprimiert. |
| Kommafrei (genetischer Code) |
Codons werden ohne Trennzeichen hintereinander gelesen. |
| Komplementationstest |
Test zur Bestimmung, ob zwei Mutationen im gleichen oder in verschiedenen Genen liegen. |
| Komplementäre Basenpaarung |
A-T (bzw. A-U), G-C. |
| Lagging Strand |
Diskontinuierlich synthetisierter DNA-Strang. |
| Leading Strand |
DNA-Strang, der während der Replikation kontinuierlich synthetisiert wird. |
| Ligase |
Verbindet DNA-Fragmente. |
| Locus |
Gen-Ort auf einem Chromosom. |
| Mediator-Komplex |
Protein-Komplex, der Transkriptionsfaktoren mit der RNA-Polymerase verbindet. |
| Meiose |
Bildung genetisch verschiedener Geschlechtszellen. |
| Mismatch-Reparatur |
Reparatur falsch gepaarter Basen nach der Replikation. |
| Mitose |
Teilung zur Bildung identischer Tochterzellen. |
| NGS |
Hochdurchsatzverfahren zur DNA-Sequenzierung. |
| NHEJ |
Nicht-homologe Endverknüpfung, schnelle aber fehleranfällige Reparatur. |
| NIPT |
Nicht-invasiver Pränataltest auf genetische Auffälligkeiten. |
| Nicht überlappend (genetischer Code) |
Jede Base gehört nur zu einem Codon. |
| Non-Disjunction |
Fehlerhafte Trennung von Chromosomen oder Chromatiden in der Meiose. |
| Nonsense-Mutation |
Mutation, die ein Stoppcodon erzeugt. |
| Nukleotid-Exzisionsreparatur |
Entfernung größerer DNA-Schäden, z. B. durch UV. |
| Okazaki-Fragmente |
Kurze DNA-Stücke des Lagging Strands. |
| Onkogen |
Gen, das das Zellwachstum fördert; Mutation kann Krebs verursachen. |
| Origin of Replication |
Startpunkt der DNA-Replikation. |
| PCR |
Methode zur Vervielfältigung von DNA. |
| Phosphorylierung |
Posttranslationale Modifikation durch Anhängen einer Phosphatgruppe. |
| Phänotyp |
Das sichtbare Merkmal, das aus dem Genotyp resultiert. |
| Pluripotent |
Stammzellen, die sich in viele Zelltypen entwickeln können. |
| Polyadenylierung |
Anhängen eines Poly-A-Schwanzes an das 3′-Ende der prä-mRNA. |
| Polygenie |
Ein Merkmal wird durch mehrere Gene beeinflusst (z. B. Hautfarbe). |
| Polymorphismus |
Natürlich vorkommende genetische Variation in einer Population. |
| Polypeptid |
Kette aus Aminosäuren, Produkt der Translation. |
| Promotor |
Startregion für Transkription. |
| Proteinbiosynthese |
Herstellung von Proteinen aus DNA-Information. |
| Präimplantationsdiagnostik |
Genetische Untersuchung von Embryonen vor der Einpflanzung. |
| RNA |
Einzelsträngige Ribonukleinsäure. |
| Redundant (genetischer Code) |
Mehrere Codons codieren für dieselbe Aminosäure. |
| Replikation |
Verdopplung der DNA. |
| Rezessiv |
Ein Allel, das nur im homozygoten Zustand zur Ausprägung kommt. |
| Rezessiver Erbgang |
Das Merkmal wird nur ausgeprägt, wenn beide Allele rezessiv sind. |
| Ribose |
Zuckerkomponente der RNA-Nukleotide. |
| Ribosom |
Zellorganelle für Proteinbiosynthese. |
| Rote Gentechnik |
Gentechnik im medizinischen Bereich (z. B. Insulinproduktion). |
| STR |
Short Tandem Repeat – sich wiederholende DNA-Sequenzen für Identitätsnachweise. |
| STR-Analyse |
Verfahren zur Identifikation mittels DNA-Wiederholungen. |
| Somatische Zellen |
Körperzellen, nicht an der Weitergabe der Erbinformation beteiligt. |
| Spaltungsregel |
Zweite Mendelsche Regel: In der F2-Generation spalten sich die Merkmale in einem bestimmten Verhältnis auf. |
| Spleißen |
Entfernung von Introns aus der prä-mRNA. |
| Spleißosom |
Komplex aus RNA und Proteinen, der Introns aus der prä-mRNA entfernt. |
| Startcodon |
Codon (meist AUG), das den Beginn der Translation markiert. |
| Stoppcodon |
Codons (UAA, UAG, UGA), die das Ende der Translation signalisieren. |
| Stoppcodon |
Beendet die Translation (z. B. UGA, UAG, UAA). |
| Synapsis |
Paarung homologer Chromosomen während der Prophase I. |
| Taq-Polymerase |
Hitzestabiles Enzym für die DNA-Synthese bei der PCR. |
| Tetrade |
Struktur aus vier Chromatiden bei der Paarung homologer Chromosomen. |
| Transkription |
Abschreiben von DNA in mRNA. |
| Transkriptionsfaktoren |
Proteine, die die Transkription regulieren. |
| Translation |
Übersetzung von mRNA in Proteine. |
| Tumorsuppressorgen |
Gen, das das Zellwachstum hemmt; Verlust kann zu unkontrolliertem Wachstum führen. |
| Ubiquitinierung |
Markierung eines Proteins für den Abbau. |
| Unabhängigkeitsregel |
Dritte Mendelsche Regel: Gene auf verschiedenen Chromosomen werden unabhängig vererbt. |
| Uniformitätsregel |
Erste Mendelsche Regel: Kreuzt man zwei reinerbige Individuen, ist die F1-Generation gleich (uniform). |
| Universell (genetischer Code) |
Der Code gilt (fast) für alle Lebewesen gleich. |
| Uracil |
Stickstoffbase in RNA, ersetzt Thymin. |
| Uracil (U) |
RNA-Base, ersetzt Thymin. |
| Weiße Gentechnik |
Gentechnik in der Industrie (z. B. Enzyme für Waschmittel). |
| X-chromosomal |
Gen liegt auf dem X-Chromosom – Erbgang betrifft meist Männer stärker. |
| Zygote |
Befruchtete Eizelle (diploid). |
| Zytokinese |
Teilung des Zellplasmas am Ende der Zellteilung. |
| iPS-Zellen |
Reprogrammierte, pluripotente Stammzellen. |
| mRNA |
Überträgt genetische Information zur Translation. |
| miRNA |
Kleine RNA, die Translation hemmen kann. |
| prä-mRNA |
Unverarbeitete mRNA direkt nach der Transkription, enthält noch Introns. |
| tRNA |
Transportiert Aminosäuren zur mRNA. |